Can we accurately assess disease activity using automated methods in large real-life MRI databases?
Résumé
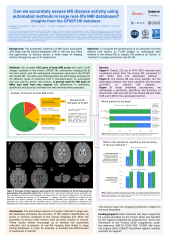
Introduction:Large real-life databases (DB) of MS patients usually consist of clinical data, including limited imaging metrics.The possibility of re-analyzing images to extract a wide range of metrics is now possible through the use of AI based methods. The presence of new lesions on longitudinal MRIs for example is used to assess the effectiveness of treatments in real-life studies. However, the automated tools that currently identify these new lesions are designed as an aid for radiologists, potentially generating false positives. The possibility of transferring these methods to analyze DB without supervision should be assessed.Objectives/Aims:To compare the performance of an automated method to classify MS patients as “active” or “inactive” based on new lesions on FLAIR images in a large real-life multicentric DB with respect to the data provided in the clinical DB.Methods:We included 1412 pairs of brain MRI scans from 868 MS patients with both FLAIR images available in the French OFSEP HD cohort imaging DB at 2 time points, and the radiological comparison captured in the clinical DB. An automated tool based on a fully convolutional neural network (trained on 159 patients) was used to detect new lesions between the corresponding longitudinal FLAIR images. Then, 160 pairs of brain MRI scans for which the automated method output and the corresponding clinical DB comparison disagree were randomly selected and their MRI were reviewed by 2 experts to constitute a ground truth. Differences in sensitivity, specificity and accuracy between the automated method and the clinical DB were assessed.Results:Overall, 222 out of 1412 (16%) intervals were considered active from the clinical DB, compared to 467 (35%) from the automated method. Over the 160 cases of disagreement included in the ground truth, the automated method correctly classified patients in 66% of the cases and the clinical DB in 34%. More specifically, the automated method was more sensitive than the clinical DB (p<0.001), but the clinical DB was more specific (p<0.001). Under simplified assumptions, we extrapolate from these results a sensitivity, specificity and accuracy of about 0.95, 0.99 and 0.92 for the clinical DB and 0.99, 0.69 and 0.96 for the automated method.Conclusion:The automated analysis of images collected in large real-life databases allows to correctly classify MS patients as active or inactive in a large majority of cases, and offers the possibility to extract other metrics such as lesion number or volume to analyze the efficacy of treatments in real-life.
Introduction:Les grandes bases de données réelles de patients atteints de SEP sont généralement constituées de données cliniques, y compris des mesures d'imagerie limitées.La possibilité de réanalyser les images pour en extraire un large éventail de mesures est désormais possible grâce à l’utilisation de méthodes basées sur l’IA. La présence de nouvelles lésions sur les IRM longitudinales est par exemple utilisée pour évaluer l'efficacité des traitements dans des études réelles. Cependant, les outils automatisés qui identifient actuellement ces nouvelles lésions sont conçus pour aider les radiologues, générant potentiellement des faux positifs. La possibilité de transférer ces méthodes pour analyser la base de données sans supervision doit être évaluée.Objectifs/Buts :Comparer les performances d'une méthode automatisée pour classer les patients atteints de SEP comme « actifs » ou « inactifs » sur la base de nouvelles lésions sur les images FLAIR dans une grande base de données multicentrique réelle par rapport aux données fournies dans la base de données clinique.Méthodes :Nous avons inclus 1412 paires d'IRM cérébrales de 868 patients atteints de SEP, avec les FLAIR disponibles aux deux points temporels dans la base de données de la cohorte OFSEP HD, et les données radiologique issues de la base de données clinique. Un outil automatisé basé sur un réseau de neurones convolutif (entraîné sur 159 patients) a été utilisé pour détecter de nouvelles lésions entre les images FLAIR longitudinales correspondantes. Ensuite, 160 paires d'IRM cérébrales pour lesquels les résultats de la méthode automatisée et la comparaison clinique correspondante de la base de données étaient en désaccord ont été sélectionnées au hasard et leur IRM a été examinée par 2 experts pour constituer une vérité terrain. Les différences de sensibilité, de spécificité et d'exactitude entre la méthode automatisée et la base de données clinique ont été évaluées.Résultats:Au total, 222 intervalles sur 1 412 (16 %) ont été considérés comme actifs à partir de la base de données clinique, contre 467 (35 %) à partir de la méthode automatisée. Sur les 160 cas de désaccord inclus dans la vérité terrain, la méthode automatisée a correctement classé les patients dans 66 % des cas et la base de données clinique dans 34 % des cas. Plus précisément, la méthode automatisée était plus sensible que la base de données clinique (p < 0,001), mais la base de données clinique était plus spécifique (p < 0,001). Sous hypothèses simplifiées, nous extrapolons de ces résultats une sensibilité, une spécificité et une précision d'environ 0,95, 0,99 et 0,92 pour la base de données clinique et 0,99, 0,69 et 0,96 pour la méthode automatisée.Conclusion:L'analyse automatisée des images collectées dans de grandes bases de données réelles permet de classer correctement les patients atteints de SEP comme actifs ou inactifs dans une grande majorité des cas, et offre la possibilité d'extraire d'autres paramètres tels que le nombre ou le volume des lésions pour analyser l'efficacité des traitements dans vrai vie.
Domaines
Informatique [cs]
Fichier principal
 Poster Comete.pdf (9.01 Mo)
Télécharger le fichier
Abstract.pdf (163.63 Ko)
Télécharger le fichier
Poster Comete.pdf (9.01 Mo)
Télécharger le fichier
Abstract.pdf (163.63 Ko)
Télécharger le fichier


